Methodik
Methodik
Der molekularbiologische Nachweis und die Bestimmung Holz-zerstörender Pilzarten verlaufen über mehrere Stufen.
In der ersten Stufe erfolgt die DNA-Isolation aus der eingesandten Probe. Abhängig von dem Material der Probe kann die Isolation eine große Herausforderung darstellen. Direkt aus befallenem Substratholz oder anorganischem Material ist die Isolation von Pilz-DNA anspruchsvoll und nicht immer erfolgreich. Die Isolation der DNA aus freiem Myzel ohne Substratholz ist in der Regel unproblematisch. Vor der Aufarbeitung wird der Probe eine synthetische DNA zugegeben. Die synthetische DNA wird bei negativen Befunden für die technische Validation der Aufarbeitung verwendet. Negativ validierte Proben werden Einsendern nicht in Rechnung gestellt.
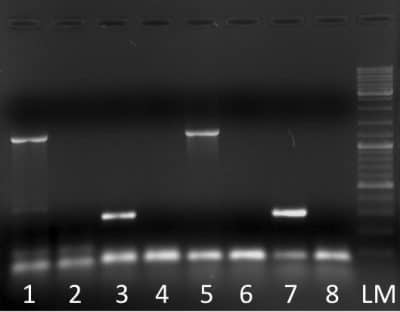
Gelelekrophorese der PCR-Produkte
Nach der DNA-Isolation erfolgt die Polymerase Kettenreaktion (PCR) mit deren Hilfe die DNA des Echten Haus-schwamms (Serpula lacrymans) eindeutig nachgewiesen werden kann. Ein erster positiver Befund wird mit einer zweiten Hausschwamm-spezifischen PCR verifiziert. Sind beide Hauschwamm-spezifischen Serpula-PCR Analysen positiv, muss zweifelsfrei davon ausgegangen werden, dass ein Befall mit Echtem Hausschwamm vorliegt.
Die PCR-Analytik erlaubt zuverlässig und objektiv, ob bei einer Probe ein Befall mit dem Echten Hausschwamm vorliegt. Das Ausmaß eines Befalls kann nur eingeschränkt mittels PCR bestimmt werden, da nur ein punktueller Ausschnitt eines Untersuchungsobjekts betrachtet wird. Dieser Aspekt unterstreicht die herausragende Bedeutung der Probenentnahme für einen aussagekräftigen Befund.
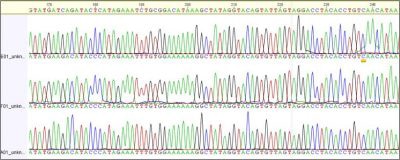
Sequenzchromogramm
Andere Holz-zerstörende Pilzarten, können mittels „PAN-PCR“ und Sequenzierung identifiziert werden. Bei diesem Ansatz wird die ITS-Region (internal transcribed spacer) der ribosomalen Gene von allen Holz-zerstörenden Pilzen mittels PCR vermehrt. Anschließend wird die DNA-Sequenz des PCR-Produktes bestimmt. Der bioinformatische Abgleich der gewonnenen Sequenzinformation mit der NCBI Datenbank (National Center for Biotechnology Information) erlaubt eine eindeutige Pilzbestimmung. Dieser universelle Ansatz führt jedoch nicht immer zu einem eindeutigen Befund. Wenn mehreren Pilzarten in einer Probe vorliegen, ergeben sich bei der Sequenzierung Überlagerungen, die eine Artbestimmung verhindern können.
Über die DNA-Isolation hinaus, unterliegen alle verwendeten Materialien und Untersuchungen einer engmaschigen Qualitätskontrolle (Spezifität und Sensitivität).
PCR
„Die PCR kann ausgehend von einem einzigen Molekül des genetischen DNA-Materials an einem Nachmittag 100 Milliarden gleichartiger Moleküle herstellen.“
— K. Mullis 1990, Scientific American
Die Polymerase-Kettenreaktion (PCR, polymerase chain reaction) erlaubt ausgehend von zwei gegenläufigen DNA-Startmolekülen in Kombination mit einer Hitze stabilen DNA-Polymerase die Vermehrung der DNA-Region zwischen den beiden Startmolekülen. Mit Hilfe der PCR kann eine Genregion innerhalb kurzer Zeit so stark vermehrt werden, dass die DNA mit dem bloßen Auge sichtbar wird.
Für die Entwicklung der PCR erhielt Karl Mullis 1993 den Nobelpreis für Chemie.
Qualitätssicherung
Die molekularbiologische Untersuchung erfolgt in Anlehnung an die Technische Regel BVL G 25.40-12013-01 zur „ PCR-Amplifikation und DNA-Sequenz analyse der 5,8S rRNA-ITS Genregion zur taxonomischen Einordnung von Pilzen“

Cleanbench Seitenansicht
Um unserem Qualitätsanspruch zu genügen, werden alle Schritte der Untersuchungen mit geeigneten Methoden kontrolliert und technisch validiert. Besonders die DNA-Isolation aus befallenem Holz erfordert eine zuverlässige technische Validation, da viele Holzinhaltsstoffe die PCR blockieren. Mit der Verwendung einer synthetischen DNA, die vor Aufarbeitung einer Probe zugesetzt wird, kann die Qualität der Aufarbeitung zuverlässig kontrolliert werden. Mit dem PCR-Nachweis dieser synthetischen DNA nach der Aufarbeitung, können hemmende Substanzen ausgeschlossen werden.
Diese Validation wird für alle Proben mit einem ansonsten negativen PCR-Befund durchgeführt. Aufgearbeitete Proben, die diese technische Validation nicht bestehen, werden dem Einsender nicht in Rechnung gestellt.
Die Spezifität der Serpula-PCR für den Echten Haus-schwamm (Serpula lacrymans) wurde gegen den Wilden Hausschwamm (Serpula himantioides), den Brauen Keller- oder Warzenschwamm (Coniophora puteana), den Weißen Porenschwamm (Antrodia vaillantii) und dem Balkenblättling (Gloeophyllum trabeum) getestet.
Die analytische Sensitivität der Serpula-PCR und der PAN-PCR liegt bei einer unteren Nachweisgrenze von 500 Kopien. Die Genome Holz zerstörender Pilze enthalten 15 – 25 Kopien der ITS-Region. Bezogen auf die Keimzahl ergibt sich für die Serpula- und PAN-PCR eine untere Nachweisgrenze von ca. 25 Pilzzellen.
Ein negativer Befund in der PCR kann das Vorhandensein von Pilz DNA unter der Nachweisgrenze nicht ausschließen.